| 2000年 9月26日 | ||||
|
||||
| オリンパス光学工業株式会社(代表取締役社長:岸本正壽)は、来たる遺伝子医療時代に向けて、がんや遺伝病などに関連する遺伝子解析を行うための高速な塩基配列計算が可能なオリゴプローブ※1設計ソフトウェアを東京大学大学院総合文化研究科の陶山明助教授と共同開発しました。本ソフトウェアは、特異性の高い塩基配列の計算が従来法※2で数週間から数ヶ月といった膨大な時間を要していたのに対し、独自のアルゴリズムを用いることにより大幅な高速化を実現し、より高精度な配列を数日で計算可能にしました。これにより、膨大な塩基配列から標的の遺伝子を高速に検出して解析するという世界的な研究課題を一気に解決する可能性があります。 |
| 現在、ヒトゲノムプロジェクトによりヒト遺伝子の約30億個もの全塩基配列が解明され、その塩基配列を医療に役立てていくポストゲノム時代に入ろうとしてます。今後、10万種以上あると言われるヒト遺伝子のうち糖尿病などの生活習慣病やがんに関連する遺伝子が明らかにされ、予防のためのリスク診断や、効果的な投薬のための薬剤応答性診断※3などの高度な遺伝子診断へ応用されることが期待されています。
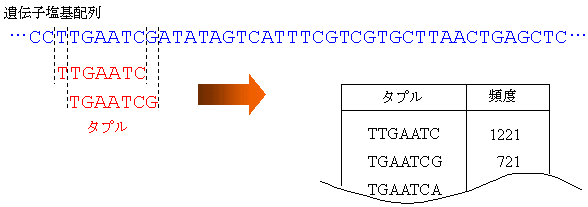
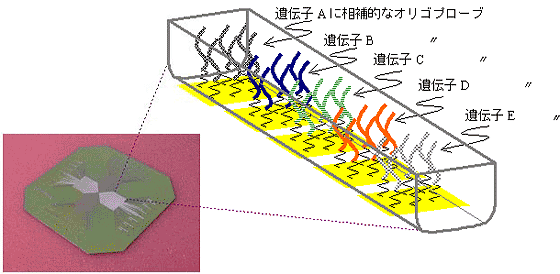
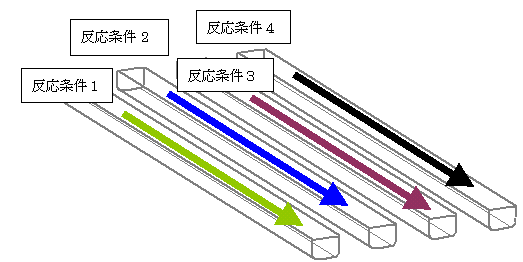
遺伝子を解析するには、組織や末梢血などから抽出したDNA※1の塩基配列を読みとります。その手法としてはDNAマイクロアレイ※4を用いたハイブリダイゼーション法※1があり、20~30塩基程度の長さのオリゴヌクレオチドプローブ(オリゴプローブ)と呼ばれる一本鎖のDNAが用いられます。このオリゴプローブは標的遺伝子に相補的な塩基配列に設計しますが、標的以外の似かよった配列の遺伝子に結合しないような配列を選択することは難しく研究者の勘や経験に頼って行っていました。このため、オリゴプローブが標的でない遺伝子と結合することがあり、結局実験で良い配列を選別するしかありませんでした。 今回開発しましたオリゴプローブ設計ソフトウェアは東京大学大学院総合文化研究科の陶山助教授のもとで考案されたタプル法※5を用い、対象の遺伝子全体を何度も走査することなく標的特異性の高いオリゴプローブ配列を設計することができます。さらにソフトウェアにそなえた計算フィルタ※6により、適当な反応温度でグループ化された、自己分子内で構造をつくらない※7理想的な塩基配列を得ることができます。これら温度特性のそろったオリゴプローブは、弊社で開発中のDNAキャピラリアレイ※8を用いれば温度条件ごとに分けて実験することができ、より正確な遺伝子診断が実現できます。 |
| 尚、「オリゴプローブ設計ソフトウェア」の技術は、9月28日に新宿・京王プラザホテルで開催されます「バイオジャパン2000」のシンポジウムで関連技術と共に発表します。 |
| 1. DNAの性質とハイブリダイゼーション法、オリゴプローブ | ||||||||||||||||||
|
||||||||||||||||||
| 2. 従来の標的特異性のチェック方法 | ||||

|
||||
| 3. 薬剤応答性診断 | |
|
|
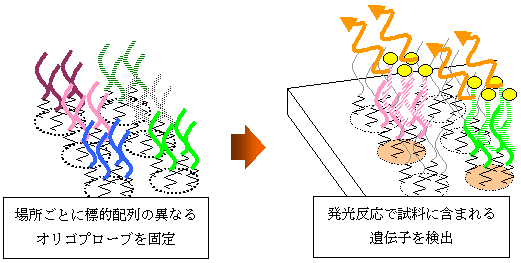
| 4. DNAマイクロアレイ | |||
|
|||
| 5. タプル法 | ||||||||
|
||||||||
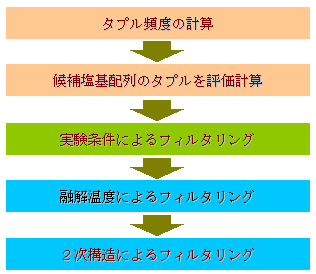
| 6. 本ソフトウェアの構成と計算フィルタ | |||
|
|||
| 7. 自己分子内構造 | |||
|
|||
| 8. 本ソフトウェアで設計したオリゴプローブのDNAキャピラリアレイでの利用 | ||||||||||
 |
||||||||||
オリンパス光学工業株式会社は、2003年10月1日をもってオリンパス株式会社と社名変更いたしました。
- 本リリースに掲載されている内容は、報道関係者向けに発表した情報です。
- 掲載内容は、発表日現在の情報であり、ご覧になっている時点で、予告なく情報が変更(生産・販売の終了、仕様、価格の変更等)されている場合があります。
- 掲載されている社名、製品名、技術名は各社の商標または登録商標です。
本文の終わりです